嘿!兄弟!我们好久不见你在哪里!嘿!朋友!如果真的是你请打招呼!哈哈哈哈哈哈哈哈哈哈哈哈好久不见大家!我回来啦!!!想我没!想我的话就多看看我啊哈哈哈哈哈哈哈哈哈!谢谢你们的喜欢和支持!!!
话不多说,开启我们今天的!GSEA!因为推测大家代码可能更常用,也更灵活方便一点,所以今天先更新代码实操部分,如果小伙伴们想要软件实操的话(应该是数据文件准备部分有点讨厌对不对,或许还有参数设置部分),可以私信我或者在群里@我!让我看看需求量如何!快让我听到你们的欢呼声!!!算了,我觉得不管大家有没有需求,我都想介绍一下,总会有人有需求的!而且我也想再熟悉一下哈哈哈哈哈哈哈哈哈! 嗨起来!开工!
0 GO/KEGG vs. GSEA
传统富集分析通常是指基于超几何分布(hypergeometric)或Fisher精确检验(Fisher's Exact Test)的方法,比如GO (Gene Ontology) 富集分析和KEGG (Kyoto Encyclopedia of Genes and Genomes) 富集分析。这些方法主要关注差异基因的表达水平,用于寻找在两组样本中,某个功能类别或生物通路中是否存在显著富集的差异基因。在这类方法中,我们通常会先设定一个显著性阈值,选取差异表达的基因,然后通过统计检验来判断这些差异基因是否在某个功能类别或通路中富集。
这些传统富集分析方法的困境在于,它往往需要我们主观地设定显著差异基因的阈值。这种固有的主观性使得这些方法对于表达变化较小的基因很难适用,或者说对于整体基因集的总体趋势缺乏全面认识。因为我们很难事先确定一个合适的阈值。这就意味着我们可能会错过那些在整体基因集中协同变化的细微但重要的信号。
GSEA的出现弥补了这一缺陷。它的独特之处在于我们不再需要预定义显著差异基因的阈值。GSEA以一种更为灵活的方式,关注整个基因集的协同变化模式(是不是有点像我们之前介绍过的看完还不会来揍/找我 | WGCNA 加权基因共表达网络分析(二)| 附完整代码 + 注释)。这意味着,即使是那些表达变化较小的基因,只要它们在整个基因集中产生了协同效应,都能够被GSEA有效地捕捉到。
当我们使用传统富集分析时,我们可能会得知某一功能通路与差异基因的相关性,但却无法深入了解整体通路的表达趋势。GSEA弥补了这一不足,它不仅提供了基因集在两种不同生物条件下的富集情况,更直观地展示了整个通路的表达趋势是上升还是下降。
我们先简单总结一下传统富集分析与GSEA的区别,方便大家心里有数地继续往下看!
传统富集分析:
- 焦点: 集中在差异基因的表达水平上,通常需要预先定义显著差异基因的阈值。
- 分析对象: 主要关注差异基因是否在特定通路或功能集中富集。
- 局限性: 受阈值设定的主观性影响,仅适用于表达变化较大的基因。
- 结果解释: 得到功能通路与差异基因的相关性,但无法回答整体通路的表达水平是上升还是下降的问题。
GSEA富集分析:
- 焦点: 从整体基因表达数据出发,关注整个基因集的协同变化。对于时间序列数据或样本有定量属性时,GSEA的优势会更明显,不需要每个分组分别进行富集,直接对整体进行处理。
时间序列数据表示了在不同时间点或条件下的基因表达变化,而具有定量属性的样本则可能包含连续的数值信息,例如疾病程度或药物剂量。
GSEA在这种情况下的优势在于,它不需要将样本分组并对每个分组单独进行富集分析。相反,GSEA可以直接对整体数据集进行处理。这意味着它能够综合考虑整个时间序列或定量属性的变化趋势,而不会将数据分割成离散的组别。
传统的富集分析可能需要将时间序列数据离散化成不同的时间点或将具有定量属性的样本分成几个组别,然后分别进行富集分析。这样的做法可能会导致信息损失和结果的不一致性,因为它无法完整地反映时间序列中或定量属性上的连续性。而GSEA通过对整个数据集的直接处理,更能够捕捉到基因集在整体上的协同变化模式,使其在处理时间序列数据或具有定量属性的样本时显得更为灵活和适用。 - 分析对象: 不局限于差异基因,能够考虑细微但协调性的变化对生物通路的影响。
- 灵活性: 无需预定义显著差异基因,适用于各种表达水平变化。
- 结果解释: 提供基因集整体的富集情况,能够回答通路的总体表达水平是上升还是下降的问题。
接下来,我们正式开始介绍G!S!E!A!!!
1 GSEA的定义
我们先来看看官网怎么说!想详细了解的小伙伴们,也可以去查看GSEA的原文:Gene set enrichment analysis: A knowledge-based approach for interpreting genome-wide expression profiles | PNAS
在此附上官网地址:https://www.gsea-msigdb.org/gsea/index.jsp
Gene Set Enrichment Analysis (GSEA) is a computational method that determines whether an a priori defined set of genes shows statistically significant, concordant differences between two biological states (e.g. phenotypes).
它说:基因集富集分析(Gene Set Enrichment Analysis, GSEA)是一种计算方法,用于确定先验定义的一组基因是否在两种生物学状态(例如表型)之间显示出具有统计学意义的一致性差异。
我们来解释一下这句话中的几个重点短语。
先验定义的一组基因:首先它是一个基因合集,它包含的是你感兴趣的基因集,比如某个通路,某个GO term或者HALLMARKER基因集等等。
两种生物学状态:也就是指实验组和对照组,比如癌症和正常、男和女等。
一致性差异:指预先定义的基因集中的基因在两种生物学状态中呈现出相似的差异状态。也就是说某个通路或GO term中的基因集在实验组和对照组张呈现出一致的上调或下调趋势。
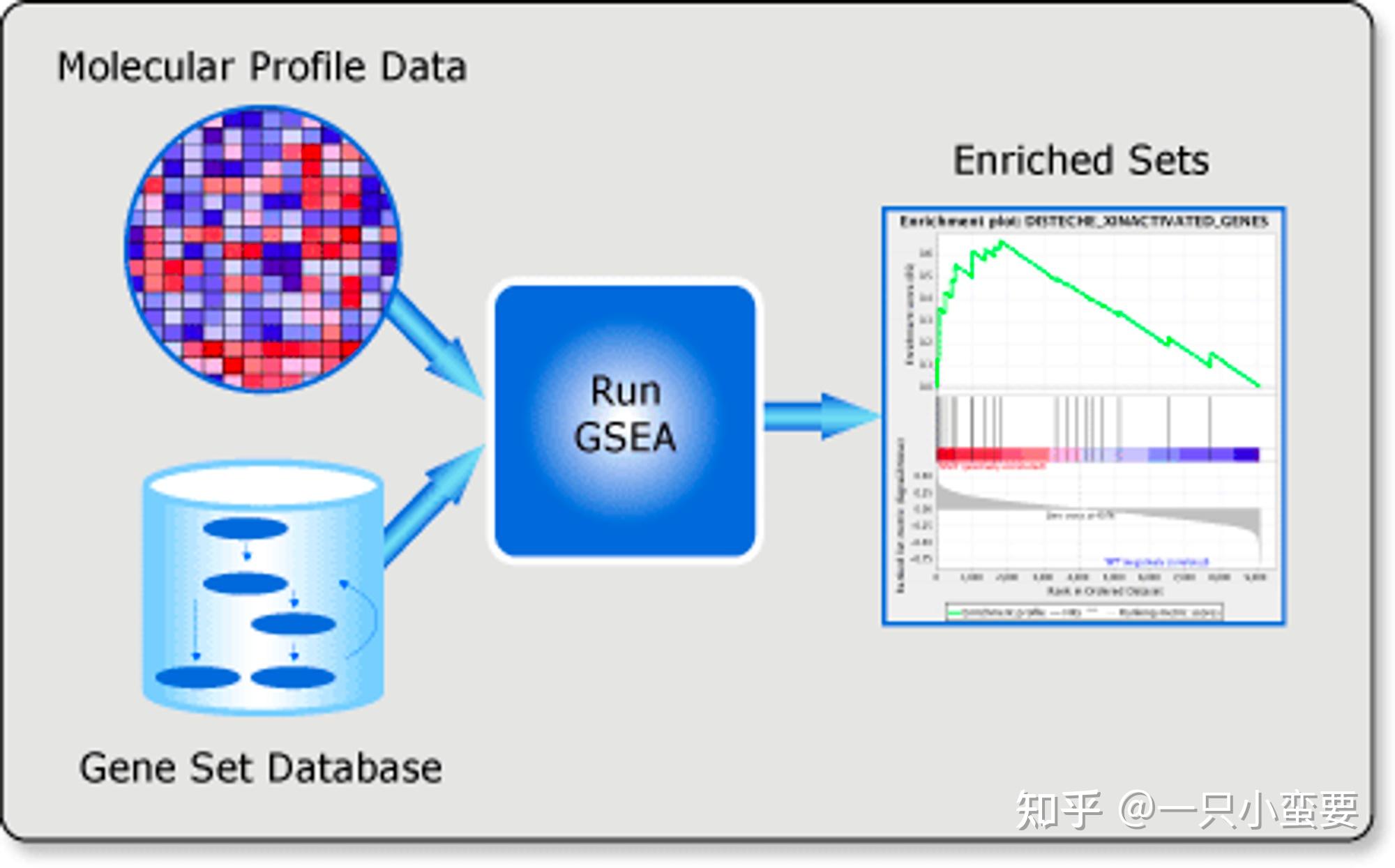
更通俗易懂地讲就是:GSEA是用来评估一个预先定义的基因集中的基因在与表型相关度排序的基因表中的分布趋势,从而判断其对表型的贡献。GSEA的输入数据包含两部分,一是已知功能的基因集(可以是GO term、MsigDB的基因集或其它符合格式的基因集定义),一是表达矩阵,软件会对基因根据其与表型的关联度(可以理解为表达值的变化)从大到小排序;也可以是我们自己排序好的基因列表,比如使用差异分析后的logFC进行排序,当然个人认为这样有点简单粗暴,可以综合考虑其他因素更好。然后判断基因集内每条注释下的基因是否富集于表型相关度排序后基因表的上部或下部,从而判断此基因集内基因的协同变化对表型变化的影响。
2 GSEA的原理
首先,咱们来看一下原理图。

给定一个排序的基因表L和一个预先定义的基因集S ,GSEA的目的是判断S里面的成员s在L里面是随机分布还是主要聚集在L的顶部或底部。这些基因排序的依据是其在不同表型状态下的表达差异,若研究的基因集S的成员显著聚集在L的顶部或底部,则说明此基因集成员对表型的差异有贡献,也是我们关注的基因集。
The gene sets are defined based on prior biological knowledge, e.g., published information about biochemical pathways or coexpression in previous experiments. The goal of GSEA is to determine whether members of a gene setStend to occur toward thetop(or bottom) of the listL, in which case the gene set is correlated with the phenotypic class distinction.
有点绕口不要怕!我们来给它变成人话!
首先我们介绍一下这几个相关的数据是啥玩意儿!
- Ranked Gene List L:该基因列表为待检测的数据集(通常是实验获得的表达矩阵文件);
- Gene Sets S:该基因集为已知功能注释的某一个通路所含基因(比如编码某个代谢通路的产物的基因、基因组上物理位置相近的基因、或者是同一GO term下的基因);
- Leading Edge Subset:核心基因集,通常为位于ES(富集得分,后面会详细介绍)的某一侧(或者Rank Gene List的某一端),它代表对富集得分贡献最大的基因成员。
所以通俗来讲,我们边看上面那张原理图边看这段话。GSEA的输入其实就是一个基因表达量矩阵,其中的样本分成了A、B两组,首先对所有基因进行排序,排序之后的基因列表其顶部可以看做是上调的差异基因(或者说在A中显著富集),其底部是下调的差异基因(或者说在B中显著富集)。GSEA分析的是一个基因集下的所有基因是否在这个排序列表的顶部或者底部富集。如果在顶部富集,该基因集(功能通路)是上调趋势,反之,如果在底部富集,则该基因集(功能通路)是下调趋势。
GSEA的关键步骤
步骤 1:计算富集得分(Enrichment Score,ES)
这个得分反映了基因集成员在排序基因列表中两端富集的程度。计算方式是从基因集所在列表的第一个基因开始,沿着列表计算一个累积统计值。每当遇到基因集中的一个基因时,增加统计值;遇到不在基因集中的基因时,则减少统计值。每一步的增减幅度与基因和表型的关联度相关。富集得分ES最后被定义为累积统计值的最大峰值。正值ES表示基因集在排序列表的顶部富集,负值ES表示基因集在底部富集。
步骤 2:评估ES的显著性
采用基于表型的排列检验(permutation test),通过对基因表达数据进行排列,重新计算基因集的ES,形成一个零分布。观察到的ES的经验P值然后相对于零分布进行计算,以确定ES的统计显著性水平。若样本数量少,也可以基于基因集做排列检验,计算p-value。
零分布指的是在进行排列检验时,对基因表达数据进行多次随机排列所得到的富集得分(ES)的分布。具体来说,对原始基因表达数据进行排列会导致基因之间的关系被破坏,生成一系列与原始数据具有相同基因数目、但基因间关系纯随机的新数据集。针对这些新数据集重新计算富集得分,得到的一系列得分形成了零分布。
ES出现的可能性指的是在这个零分布中,观察到的富集得分(ES)在零分布中的相对位置,或者说在这个分布中有多少比例的随机排列数据得到的ES值超过或等于观察到的ES。这样的比例就反映了观察到的ES相对于纯随机情况的显著性水平。
在基于表型的排列检验中,通过与零分布比较,可以计算观察到的ES相对于纯随机排列的偶然性。如果观察到的ES远远高于零分布中的大多数值,那么可以认为这个ES在统计上是显著的,反之则可能是由于随机性而无法区分。因此,零分布的建立和比较为我们提供了一种基于随机性的统计检验手段,用于评估富集分析结果的显著性。
步骤 3:多重假设检验矫正
在考虑整个基因集数据库时,进行多重假设检验矫正。首先,对每个基因集的ES进行标准化,得到归一化富集得分(Normalized Enrichment Score,NES),以考虑基因集的大小。然后,通过计算每个NES对应的错误发现率(False Discovery Rate,FDR),来控制误报的比例。这一步骤确保在整个分析中维持统计显著性的准确性,避免因多次检验而引入的假阳性。计算NES也有另外一种方法,是计算出的ES除以排列检验得到的所有ES的平均值。(注:GSEA采用 p-value < 5%,q-value < 25% 进行数据过滤。)
所以总结一下,GSEA的基本步骤就是:
- 输入所有基因表达量矩阵,样本分为两类;
- 将基因按照表达与分类的相关性进行排序;
- 计算富集得分(Enrichment Score,ES);
- 估计ES的显著性水平(P值);
- 多重假设检验校正(FDR值)。
GSEA的最终目的就是确定预先定义的基因集是否随机分布在排序的基因列表中。
GSEA软件默认的输入是基因集合(基因表达量矩阵和样本分组),然后内置的进行归一化,进行差异分析,计算Signal2Noise(默认)等统计量,其本质就是进行差异分析,计算出类似logFC的统计值,但,GSEA的归一化算法是否适用于我们输入的表达量矩阵,在计算基因的logFC时有没有考虑生物学重复本身的变化程度,这些都有可能导致其计算出的值并不一定能满足我们的需求,更加有效的做法是采用专用的差异分析工具(比如DESeq2、edgeR、limma等)计算出的logFC值来进行分析。
需要差异分析保姆级教程的小伙伴们,请查看:看完还不会来揍/找我 | 差异分析三巨头 —— DESeq2、edgeR 和 limma 包 | 附完整代码 + 注释 有进阶需求的小伙伴们还可以再看看这个:熊读文献|别再用DEseq2和edgeR进行大样本差异表达基因分析了
GSEA的核心是富集得分(Enrichment Score,ES)的计算,除了GSEA软件外,还有很多的工具也都支持这个算法,如果想要利用DESeq2等工具自定义计算出的基因排序列表进行富集分析,其实更推荐使用clusterProfiler等第三方工具哟!
咱们今天就是要给大家演示如何使用clusterProfiler进行GSEA富集分析!
下面开始!
3 代码实战
今天呢,我们就从差异分析后的数据,也就是已经获得排序信息的数据开始,直接进行GSEA富集分析!数据为使用拉帕替尼治疗前后的基因表达谱经过limma包处理后的差异分析结果,我们以logFC作为排序依据进行后续的GSEA富集分析。
老样子!本文所用到的数据和代码,我已经上传到了GitHub,有需要的话,大家可以在公众号后台回复GSEA即可获得存放数据的链接,很多需要注意的问题也会在代码注释中进行详细说明。不过我在分享过程中也会把每一步的输入数据和输出结果进行展示,大家可以作为参考并调整自己的数据格式,然后直接用自己的数据跑,也是没有任何问题的!
############################### GSEA 富集分析 ################################### 加载GSEA分析所需要的包library(clusterProfiler) # GSEA富集/基因集读取# 导入差异分析后的数据,以便后续使用logFC进行基因排序load("./gsea_data/DEG_limma.Rdata")head(DEG_limma)# logFC AveExpr t P.Value adj.P.Val B# ELOVL6 1.0386031 3.382675 80.14137 1.250078e-08 0.0002957671 9.555464# PAICS 1.0957881 4.859392 74.97349 1.715935e-08 0.0002957671 9.432400# DSCC1 1.0066575 3.714065 64.41613 3.529092e-08 0.0003055031 9.109090# SLBP 0.7234739 5.742791 64.32998 3.551600e-08 0.0003055031 9.105962# CDKN2B -2.0770246 4.617310 -59.21432 5.264576e-08 0.0003055031 8.902549# CDKN3 0.8263725 4.852420 58.56735 5.546570e-08 0.0003055031 8.874130# 加载基因集,基因集介绍往下滑!geneSet_go <- read.gmt("./gsea_data/c5.go.bp.v2023.2.Hs.symbols.gmt")head(geneSet_go)# term gene# 1 GOBP_10_FORMYLTETRAHYDROFOLATE_METABOLIC_PROCESS AASDHPPT# 2 GOBP_10_FORMYLTETRAHYDROFOLATE_METABOLIC_PROCESS ALDH1L1# 3 GOBP_10_FORMYLTETRAHYDROFOLATE_METABOLIC_PROCESS ALDH1L2# 4 GOBP_10_FORMYLTETRAHYDROFOLATE_METABOLIC_PROCESS MTHFD1# 5 GOBP_10_FORMYLTETRAHYDROFOLATE_METABOLIC_PROCESS MTHFD1L# 6 GOBP_10_FORMYLTETRAHYDROFOLATE_METABOLIC_PROCESS MTHFD2L# 这里展示的是GO的基因集# 可以看到共两列,第一列为term,可以理解为通路名称,第二列为基因MSigDB数据库介绍
这里我们介绍一下GSEA官方给出的一些预先定义的基因集,也就是GSEA | MSigDB (https://www.gsea-msigdb.org/gsea/msigdb/index.jsp)。
MSigDB(Molecular Signatures Database)是一个汇集了经过良好注释的基因集合的数据库,被广泛用于分析基因富集通路。在MSigDB的官网上,我们可以通过关键字搜索基因集,按名称或集合浏览基因集,查看基因集及其注释,下载基因集,计算我们提供的基因集与MSigDB中的基因集之间的重叠,按基因家族对基因集的成员进行分类,以及在提供的公共表达概要中查看基因组的表达谱。不仅如此,我们还可以调查在线生物网络存储库NDEx中的基因集。
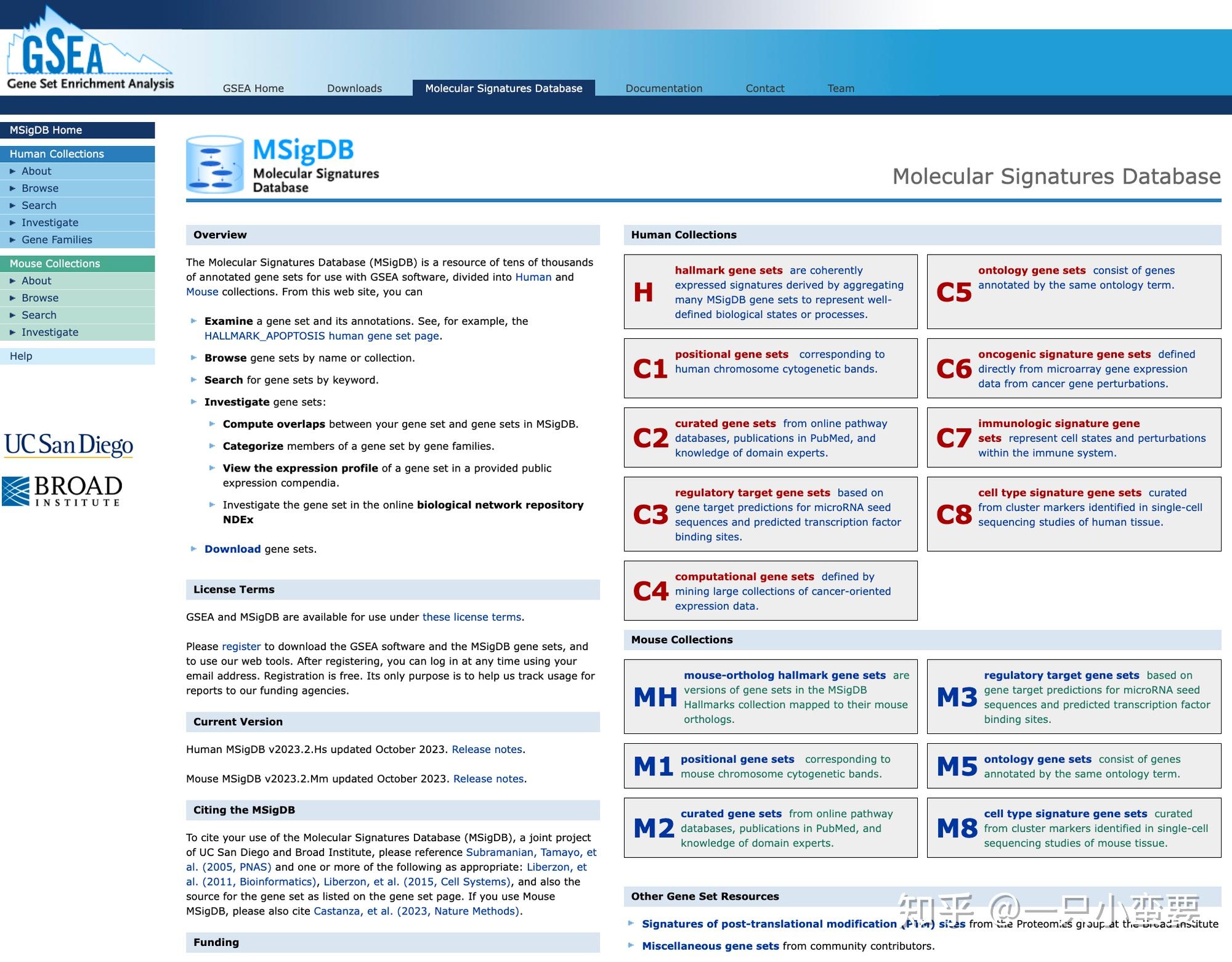
我们可以看到它包含人类和小鼠的基因集,数据库一直处于更新中,最新版本就像上图展示的那样,我们今天就先重点介绍人类基因集,它们被划分为9个大类,包括H(hallmarker gene sets)、C1(positional gene sets)、C2(curated gene sets)等等。我们接下来就具体介绍一下这9大基因集,方便大家判断自己的数据更适合使用哪些基因集进行富集分析(当然也可以自定义基因集,也就是新发现的基因集或其它感兴趣的基因的集合,甚至有些物种没有现成的基因集,我们只能自制,所以在之后我会教大家怎么制作自定义基因集,有需求的小伙伴们也可以催催我哈哈哈哈哈哈哈哈哈哈哈哈)。
MSigDB的当前最新版本大家可以在https://docs.gsea-msigdb.org/#MSigDB/Release_Notes/MSigDB_2023.2.Hs/进行查看。
- H(hallmarker gene sets): Hallmark基因集总结并代表了特定的明确定义的生物状态或过程,它包含由多个已知的基因集构成的超级基因集,每个H类别的基因集都对应多个基础的其他类别的基因集,较为常用。我们可以详细查看一下!按照下面的步骤依次点进去!
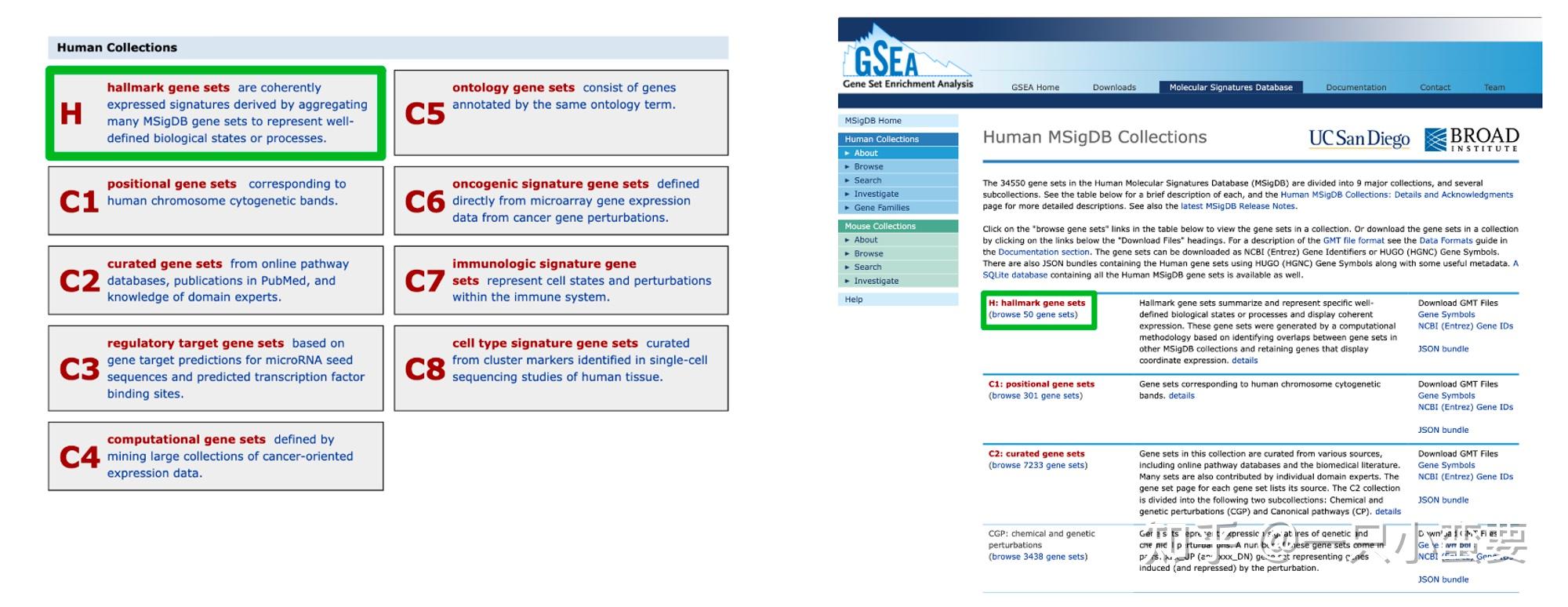

我们以第一个脂肪生成为例,点击后会出现如下界面,里面会包含这个基因集的简介、所包含的相关的基因集信息、以及与此基因集相关的200个基因信息等等。

后面的几个大类我就不这么一个一个点进去了哈,大家自行操作!
2. C1(positional gene sets): 与基因在染色体上的位置相关的基因集合,根据不同染色体编号进行二级分类,不太常用,感觉性别相关研究可能会用到。
3. C2(curated gene sets): 包含已知数据库、文献和专家支持的基因集信息,每个基因集的基因集页面会列出其来源。C2集合分为以下两个子集合:化学和遗传扰动 (CGP,chemical and genetic pertubations) 和规范通路 (CP,canonical pathways)。

4. C3(regulatory target gene sets): 代表转录因子或microRNA调控潜在靶标的基因集。些集合由按它们在非蛋白质编码区域中共享的元素分组的基因组成。这些元件代表启动子和3'-UTR中已知或可能的顺式调节元件。C3集合分为两个子集合:microRNA靶基因(MIR,microRNA targets)和转录因子靶基因(all transcription factor targets)。

5. C4(computational gene sets): 通过挖掘大量面向癌症的表达数据来定义的计算基因集。C4集合现阶段(它更新蛮快的嘛!上次看才俩!)分为三个子集合:3CA(Curated Cancer Cell Atlas)、CGN(cancer gene neighborhoods)和CM(cancer modules)。
6. C5(ontology gene sets): 包含由相同本体术语注释的基因的基因集。C5集合分为两个子集合,第一个来自基因本体资源(GO,Gene Ontology),其中包含BP、CC和MF组件;第二个来自人类表型本体(HPO,Human Phenotype Ontology)。
7. C6(oncogenic signature gene sets): 代表通常在癌症中失调的细胞通路特征的基因集。大多数特征直接来自 NCBI GEO 的微阵列数据或来自内部未发表的涉及已知癌症基因扰动的分析实验。
8. C7(immunologic signature gene sets): 代表免疫系统内细胞状态和扰动的基因集。
9. C8(cell type signature gene sets):包含针对人类组织单细胞测序研究中确定的细胞类型的精选簇标记的基因集。
以上就是关于这几个基因集的介绍啦!下载使用的话需要注册账号,很简单的!不要担心!
那我们就以C6为例向大家介绍基因集的具体下载过程!
首先我们进入官网后,依次按照下图所示方式操作即可!


可以下载整个基因集,也可以下载单个通路的基因集进行分析。一般我们最常用的就是gmt格式,还有图中可以看到,有Gene Symbols、NCBI (Entrez) Gene IDs等等,大家要依据自己的数据情况进行选择。从上面的代码中展示的数据内容大家可以知道,我们今天下载使用的是Gene Symbols。那MSigDB数据库就介绍完毕啦,咱们继续往后看!
代码实战正式开始
为了避免大家还得往上滑,我把之前的内容复制了一次又哈哈哈哈哈哈哈哈哈。
############################### GSEA 富集分析 ################################### 加载GSEA分析所需要的包library(clusterProfiler) # GSEA富集/基因集读取# 导入差异分析后的数据,以便后续使用logFC进行基因排序load("./gsea_data/DEG_limma.Rdata")head(DEG_limma)# logFC AveExpr t P.Value adj.P.Val B# ELOVL6 1.0386031 3.382675 80.14137 1.250078e-08 0.0002957671 9.555464# PAICS 1.0957881 4.859392 74.97349 1.715935e-08 0.0002957671 9.432400# DSCC1 1.0066575 3.714065 64.41613 3.529092e-08 0.0003055031 9.109090# SLBP 0.7234739 5.742791 64.32998 3.551600e-08 0.0003055031 9.105962# CDKN2B -2.0770246 4.617310 -59.21432 5.264576e-08 0.0003055031 8.902549# CDKN3 0.8263725 4.852420 58.56735 5.546570e-08 0.0003055031 8.874130# 加载基因集,基因集介绍往下滑!geneSet_go <- read.gmt("./gsea_data/c5.go.bp.v2023.2.Hs.symbols.gmt")head(geneSet_go)# term gene# 1 GOBP_10_FORMYLTETRAHYDROFOLATE_METABOLIC_PROCESS AASDHPPT# 2 GOBP_10_FORMYLTETRAHYDROFOLATE_METABOLIC_PROCESS ALDH1L1# 3 GOBP_10_FORMYLTETRAHYDROFOLATE_METABOLIC_PROCESS ALDH1L2# 4 GOBP_10_FORMYLTETRAHYDROFOLATE_METABOLIC_PROCESS MTHFD1# 5 GOBP_10_FORMYLTETRAHYDROFOLATE_METABOLIC_PROCESS MTHFD1L# 6 GOBP_10_FORMYLTETRAHYDROFOLATE_METABOLIC_PROCESS MTHFD2L# 这里展示的是GO的基因集# 可以看到共两列,第一列为term,可以理解为通路名称,第二列为基因# 我们再展示一个基因集,它来自C6,geneSet_onco <- read.gmt("./gsea_data/c6.all.v2023.2.Hs.symbols.gmt")head(geneSet_onco)# term gene# 1 AKT_UP.V1_DN ACKR3# 2 AKT_UP.V1_DN ADGRL1# 3 AKT_UP.V1_DN ADHFE1# 4 AKT_UP.V1_DN ALPL# 5 AKT_UP.V1_DN AMPD2# 6 AKT_UP.V1_DN ANGPTL4# 接下来我们进行基因排序geneList <- DEG_limma$logFC # 获取GeneListnames(geneList) <- rownames(DEG_limma) # 对GeneList命名geneList <- sort(geneList, decreasing = T) # 从高到低排序# 排序后的基因列表head(geneList)# ETV4 MYEOV ETV5 DUSP6 FGG FOSL1 # 2.893571 2.789140 2.617158 2.500177 2.442258 2.355249# 开始GSEA富集分析GSEA_enrichment <- GSEA(geneList, # 排序后的gene TERM2GENE = geneSet_onco, # 基因集 pvalueCutoff = 0.05, # P值阈值 minGSSize = 20, # 最小基因数量 maxGSSize = 1000, # 最大基因数量 eps = 0, # P值边界 pAdjustMethod = "BH") # 校正P值的计算方法result <- data.frame(GSEA_enrichment)dim(GSEA_enrichment@result)# [1] 114 11# 可以看到,最终富集到114条通路,结果有11列,咱们来看看每列都是什么吧!GSEA富集结果展示:

咱们解读一下每一列:
ID: 这一列代表基因集的标识符或名称。Description: 这一列表示基因集的描述或注释,通常是对基因集功能、通路或生物学过程的解释。setSize: 这一列表示基因集中包含的基因数量。enrichmentScore: 这一列表示富集得分(Enrichment Score),它是一个反映基因集在基因表达数据中富集程度的统计量。NES: 这一列表示标准化富集得分(Normalized Enrichment Score),它是将富集得分标准化后的值,使得不同基因集的富集得分可比较。pvalue: 这一列表示富集得分的显著性水平(p-value),用于衡量基因集在基因表达数据中的显著性富集。p.adjust: 这一列表示多重比较校正后的p-value,通常使用FDR或其他方法进行校正。qvalue: 这一列表示估计的FDR(False Discovery Rate),用于控制多重假设检验引起的假阳性。rank: 这一列表示基因集在排序后的基因列表中的排名。leading_edge: 这一列指示哪些基因在计算富集得分时对富集结果产生了主要贡献。- tags=60%: 这表示60%的基因集中的基因在富集分析中对结果产生了影响,tags表示核心基因在该基因集中在基因总数的占比。
- list=10%: 这表示在富集分析中使用的整体基因集中,有10%的基因在该基因集中,list表示核心基因占所有基因总数的比例。
- signal=55%: 这表示在整个基因集中,有55%的基因在样本中显示出富集信号,即在表达数据中呈现出差异表达的特征(由tags和list计算得到)。
core_enrichment: 这一列指示哪些基因是核心富集基因,对于形成富集得分起关键作用。
# 接下来,我们进行富集结果可视化# 加载必要包library(enrichplot) # 富集结果可视化# 特定通路作图——单个通路gseaplot2(GSEA_enrichment, "KRAS.600.LUNG.BREAST_UP.V1_DN", color = "red3", pvalue_table = T)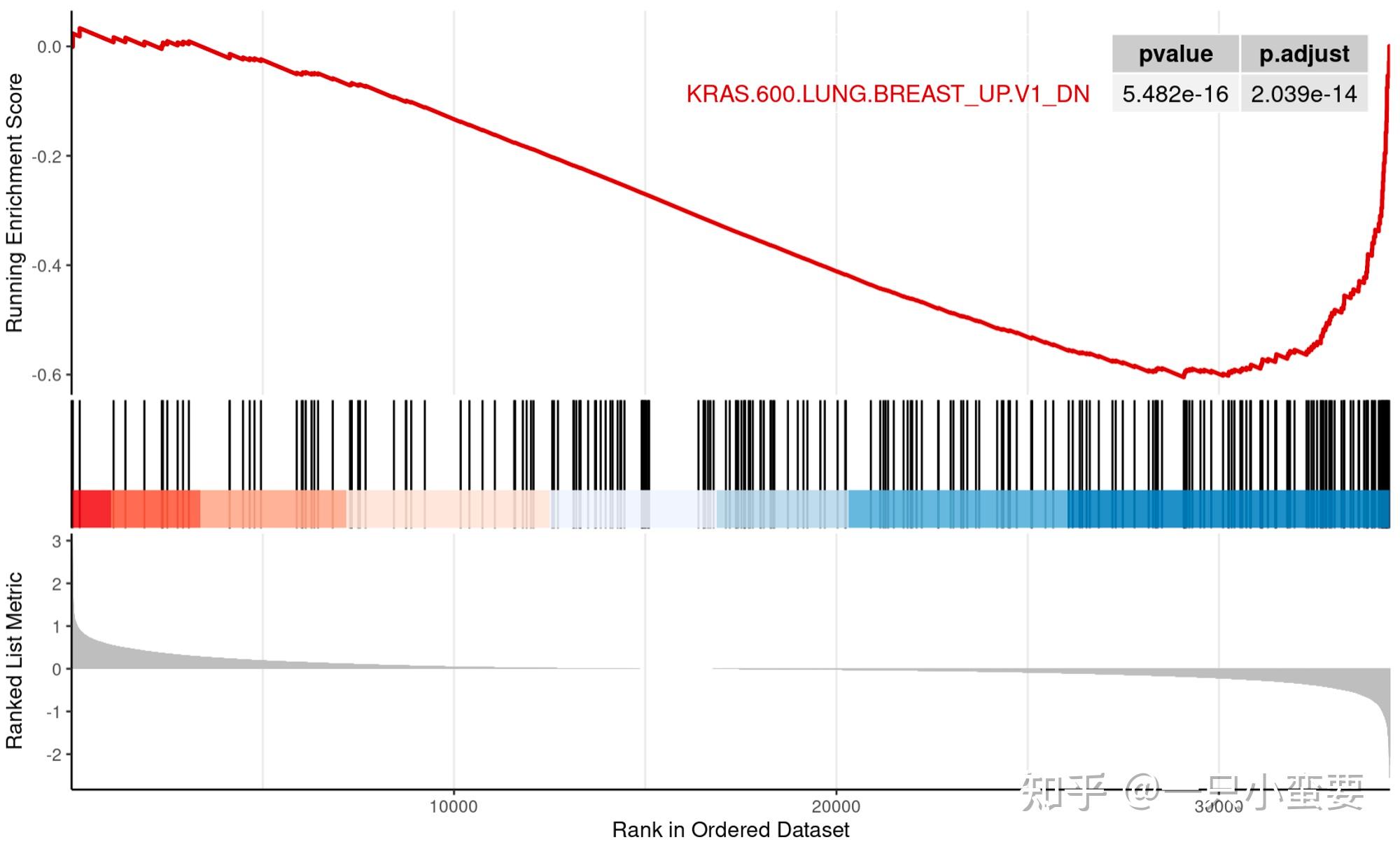
我们重点解读一下这个图图!
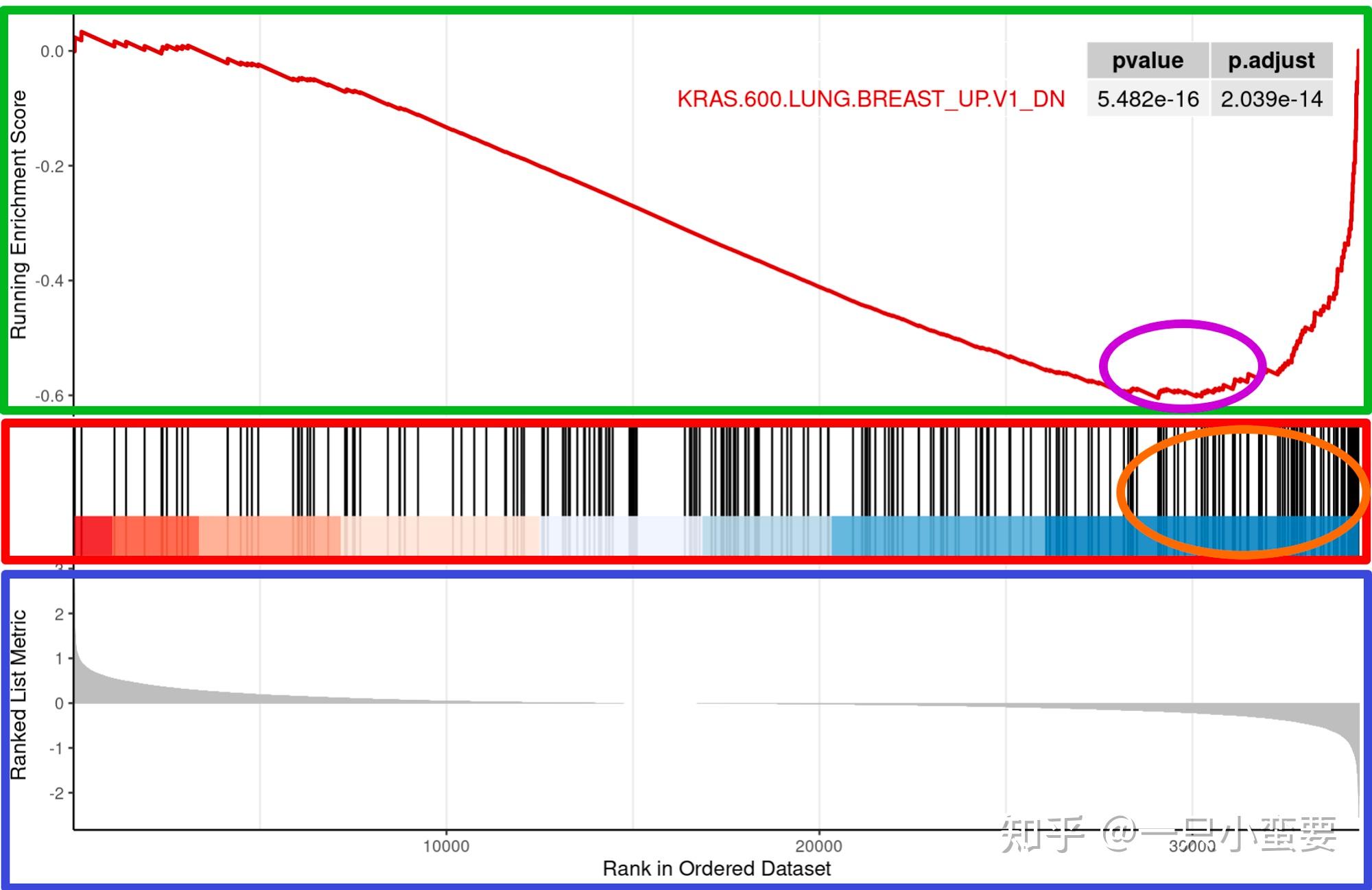
- 富集得分(ES) 折线图:该图最上部的绿色框框部分展示了ES(富集得分)的计算过程。从左至右每到一个基因,都计算出一个ES值,连接起来形成折线。紫色圈圈所在的位置,也就是最高峰处(左侧或右侧)的ES值则表示这个基因集的富集得分,即在基因列表中的某种排序下,这个基因集的显著性富集程度。
- 基因位置线条图:中间红色框框部分的线条图表示基因集中每个基因在整个基因列表中的排序位置,每一条线代表基因集中的一个基因。
- 基因与表型关联矩阵:最下面的蓝色框框部分展示了基因与表型的关联矩阵,是排序后所有基因的排序值的分布,以灰色蝴蝶图进行展示(同样的样本得到的结果中所有通路图的这部分都是一样滴),表示基因顺序和排序得分之间的正相关和负相关的关系。红色表示与第一个表型正相关,也就是指红色部分对应的基因在表型一(比如给药组)中表达较高;蓝色表示与第二个表型正相关,也就是指蓝色部分对应的基因在表型二(比如第对照组)中表达较高。
至于左右分别对应哪个表型,其实取决于你在前期获取基因排序时的处理方式(比如差异基因分析指定对比组别时的设定),一般是左边为实验组,右边为对照组,当然不一样也很正常!这个部分的话,使用软件分析就会更清晰明了,因为它会明确标准每一侧代表的表型。(咱就是说有点羡慕是不是!像下面这张图展示的这样:

4. Leading-edge subset:Leading-edge subset是对富集得分贡献最大的基因成员的集合,通常是最高峰对应的那部分基因,即核心基因集,也就是图中橙色圈圈部分。通常情况下,如果ES为正值,则是峰左侧的基因;如果ES为负值,则是峰右侧的基因。这部分基因在富集分析中对整体结果贡献最显著。
5. 结果显著性判定标准:对于分析结果中,我们一般认为|NES| > 1、p-val < 0.05、FDR q-val < 0.25的通路是显著富集的。NES的绝对值越大,FDR值就越小,说明分析的结果可信度越高。p-val是针对某一功能基因集得到的ES值的统计显著性,P值越小,说明基因的富集性越好,但P值很小时,FDR值也可能很大,这说明和其他功能基因子相比较,它的富集并不是很显著,原因可能是数据样本量较少、杂交信号微弱或者是选择的功能基因子集并未很好得反映样本的物理学意义。但如果样本数量过少,而且选择了gene_set作为Permutation type,则需要使用更为严格的标准,比如FDR < 0.05。
我们还可以再一张图中展示多个通路!
# 特定通路绘图——多个通路gseaplot2(GSEA_enrichment, c("VEGF_A_UP.V1_UP", "VEGF_A_UP.V1_DN"), color = c("red3", "blue4"), pvalue_table = T)
也可以直接展示富集到的通路!
# 展示富集到的通路,我们这里选择展示前15个dotplot(GSEA_enrichment, showCategory = 15, color = "p.adjust")
我们还可以将激活和抑制类通路(这个说法不太合适其实,咱们之后聊聊)分别进行展示!
# 将通路分为激活和抑制两个部分进行展示library(ggplot2) # 画图图dotplot(GSEA_enrichment, showCategory = 10, split = ".sign") + facet_grid(~.sign) + theme(plot.title = element_text(size = 10, color = "black", hjust = 0.5), axis.title = element_text(size = 10,color = "black"), axis.text = element_text(size = 10,color = "black"), axis.text.x = element_text(angle = 0, hjust = 1 ), legend.position = "right", legend.text = element_text(size = 10), legend.title = element_text(size = 10))
其实软件可以直接得到更多的结果,不过感觉其实很多都是用不到的,最常用的还是前面那个通路图,不过我们也会在后面的软件实操介绍中对各个图进行详细讲解的!
那今天就先到这里啦!这些图都是内置函数画出来的,咱们以后会陆续更新更漂亮的图图画法!期待!
要练习
之前小伙伴建议在每次的代码实战介绍结束后,可以进一步提供练习数据,可以帮助大家熟悉学到的东西,俺觉得超级有理!这不!要练习模块正式开始咯!给大家提供了一份基因经过Signal2Noise排序的数据(也就是软件默认的排序方法),同时提供了表达数据(如果有小伙伴想从差异分析开始练习,那俺必须支持呐!)。老办法!大家可以在后台回复GSEA获取练习数据。在这里给大家先展示一下数据内容!大家按照SCORE列进行排序后即可进行富集分析,选择自己认为合适的基因集就好啦!(好像这次的有点简单,那大家顺带解读一下结果好不好!)
数据介绍:基因表达数据来自GEO数据库GSE175906。该数据集由六个样本组成,分为两组,每组三个样本。这些样本代表了拉帕替尼治疗前后的基因表达谱。大家可以使用R包limma进行差异基因分析,然后采用GSEA富集分析方法对信号通路进行探究,也可以直接使用已经进行Signal2Noise排序的数据进行GSEA富集分析。
rank_Signal2Noise <- readRDS("./gsea_data/rank_Signal2Noise.rds")head(rank_Signal2Noise)# # A tibble: 6 × 3# NAME TITLE SCORE# <chr> <chr> <dbl># 1 SLC15A2 solute carrier family 15 member 2 [Source:HGNC Symbol;Acc:HGNC:10921] 4.46# 2 MUC19 mucin 19, oligomeric [Source:HGNC Symbol;Acc:HGNC:14362] 4.32# 3 ELSPBP1 epididymal sperm binding protein 1 [Source:HGNC Symbol;Acc:HGNC:14417] 4.29# 4 AGXT alanine--glyoxylate aminotransferase [Source:HGNC Symbol;Acc:HGNC:341] 4.28# 5 CYP4F8 cytochrome P450 family 4 subfamily F member 8 [Source:HGNC Symbol;Acc:HGNC:2648] 3.91# 6 MMP13 matrix metallopeptidase 13 [Source:HGNC Symbol;Acc:HGNC:7159] 3.80需要差异分析保姆级教程的小伙伴们,请查看:看完还不会来揍/找我 | 差异分析三巨头 —— DESeq2、edgeR 和 limma 包 | 附完整代码 + 注释 有进阶需求的小伙伴们还可以再看看这个:熊读文献|别再用DEseq2和edgeR进行大样本差异表达基因分析了
咦,你们说咱们有没有必要建立一个要练习的交流群!大家可以把练习得到的结果或者遇到的问题或者学到的经验进行交流分享!有趣!小伙伴们可以在后台私信或咱们的生信小白要知道交流群(大家可以在公众号主页作者精选模块看到交流群相关信息)或者这里!提出建议!蟹蟹!!!
结尾碎碎念
关于基因列表到底该如何排序,或许有我们机智的小伙伴们愿意看看这里:Ranking metrics in gene set enrichment analysis: do they matter? | BMC Bioinformatics
它的结论是:
The absolute value of Moderated Welch Test has the best overall sensitivity and Minimum Significant Difference has the best overall specificity of gene set analysis.
嗯……对!大家,冲啊!
参考资料
- https://blog.csdn.net/nixiang_888/article/details/107062461
- https://cloud.tencent.com/developer/article/1426130
- https://www.bilibili.com/video/BV1fA411V7kR
- http://www.bio-info-trainee.com/4023.html
- https://zhuanlan.zhihu.com/p/347148653
- https://zhuanlan.zhihu.com/p/479856659
- https://mp.weixin.qq.com/s/LLODJwvEbcwuBTLJEEpTxA
- http://blog.genesino.com/2017/10/gsea/#msigdb
- https://zhuanlan.zhihu.com/p/44091458